Nuevo mecanismo molecular en la reparación de ADN
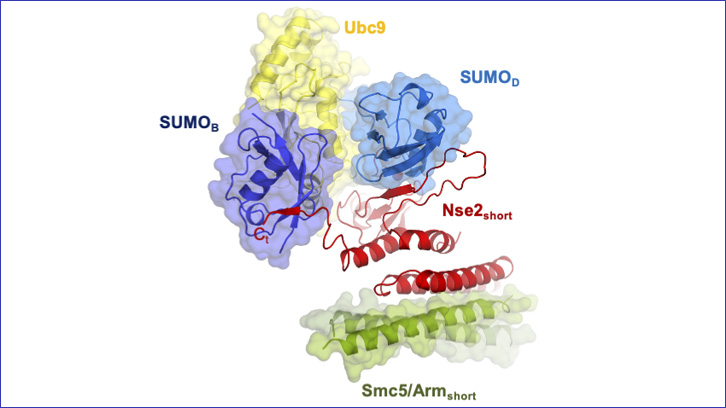
Un estudio liderado por un equipo de investigación del IBB y del Departamento de Bioquímica y Biología Molecular de la UAB ha revelado el funcionamiento de un complejo enzimático formado por un conjunto de proteínas: una E3 ligasa, unida a una E2 y a dos SUMO, y ha identificado los puntos clave para el correcto funcionamiento de esta E3 ligasa, que participa en la reparación de daño en el ADN a través de modificaciones post-traduccionales por SUMO.
Las modificaciones post-traduccionales de las proteínas son aquellos cambios que se realizan una vez la proteína ya ha sido sintetizada por el ribosoma. Estas modificaciones tienen mucha importancia, puesto que pueden producir cambios en la actividad biológica de las proteínas modificadas, generando así un importante mecanismo de regulación celular.
SUMO (Small Ubiquitin-like MOdifier) es una proteína pequeña que tiene la capacidad de unirse a otras proteínas a través de estas modificaciones post-traduccionales, dando lugar al proceso llamado “SUMOilación”. Hay un gran número de proteínas implicadas en la SUMOilación, necesarias por la propia la activación de SUMO y también sujetas a esta modificación post-traduccional. Entre ellas destaca el complejo Smc5/6, un complejo formado por diversas proteínas que participa activamente en la reparación del daño en el ADN.
En este artículo, presentamos la estructura tridimensional de dos proteínas del complejo Smc5/6: Nse2 y Smc5, interaccionando con dos proteínas clave en el proceso de SUMOilación: E2 y la propia proteína SUMO unidas por un enlace tioéster. Gracias a la observación de este complejo enzimático, hemos podido definir los puntos clave de la unión entre estas proteínas que nos han permitido caracterizar el mecanismo de la actividad SUMO E3 ligasa de Nse2 y su implicación en la reparación del ADN.
Este trabajo ha sido realizado por el grupo de investigación dirigido por el Dr. David Reverter, vinculado al Institut de Biotecnologia i Biomedicina y al departamento de Bioquímica y Biología Molecular de la UAB, en colaboración con el grupo del Dr. Jordi Torres-Rosell, vinculado al IRB-Lleida. Las primeras autoras del artículo somos la Dra. Nathalia Varejão y la investigadora predoctoral Jara Lascorz, también pertenecientes al IBB-UAB.
Instituto de Biotecnología y Biomedicina
Departamento de Bioquímica y Biología Molecular
Universidad Autónoma de Barcelona
Referencias
Varejão, N., Lascorz, J., Codina-Fabra, J., Bellí, G., Borràs-Gas, H., Torres-Rosell, J., & Reverter, D. (2021). Structural basis for the E3 ligase activity enhancement of yeast Nse2 by SUMO-interacting motifs. Nature Communications, 12(1), 7013. https://doi.org/10.1038/s41467-021-27301-9


