Mutagénesis y transferencia lateral de genes: procesos implicados en la defensa bacteriana frente a los bacteriófagos
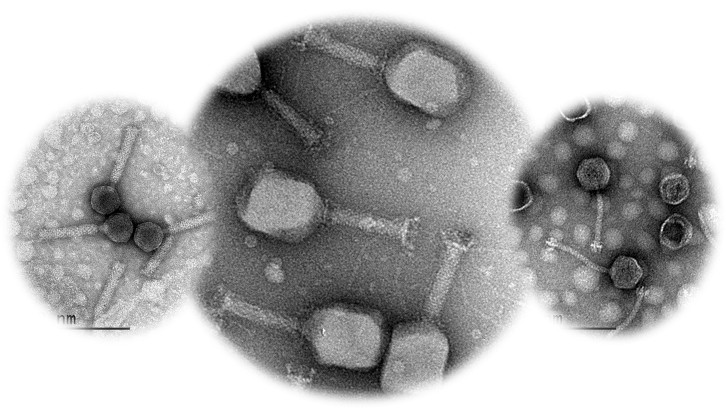
Uno de los mayores retos sanitarios de la actualidad es combatir la resistencia bacteriana a los antibióticos. El uso de bacteriófagos (virus que únicamente infectan a bacterias) es una de las estrategias más prometedoras para tal fin, pero las bacterias también pueden desarrollar resistencias y mecanismos de defensa contra ellos. El grupo de Microbiología Molecular del Departamento de Genética y de Microbiología estudia cómo emergen estas resistencias y mecanismos de defensa y ha obtenido resultados destacados en un trabajo donde se ha usado un cóctel de tres bacteriófagos contra la bacteria Salmonella enterica serovar Typhimurium, uno de los principales agentes causantes de enfermedad gastrointestinal en humanos.
La resistencia bacteriana a los antibióticos supone el mayor problema sanitario del siglo XXI y urge desarrollar nuevas estrategias para abordarlo de forma global desde una perspectiva One Health. La terapia fágica ha ido ganado especial relevancia ante esta preocupante realidad. No obstante, el uso de fagos en el control de patógenos puede generar la emergencia de variantes bacterianas resistentes a ellos al igual a lo que ha ocurrido con la emergencia de resistencia a antibióticos.
El grupo de Microbiología Molecular del Departamento de Genética y Microbiología de la UAB, dirigido por la Dra. Montserrat Llagostera, ha estudiado la emergencia de resistencias y de mecanismos de defensa frente a un cóctel de tres bacteriófagos del patógeno Salmonella enterica serovar Typhimurium. Para ello, se han aislado y estudiado, mediante métodos de microbiología convencional, molecular y de bioinformática, variantes de Salmonella con susceptibilidad reducida a los fagos en cultivos de laboratorio de Salmonella infectados con fagos (LAB), en lonchas de jamón cocido contaminadas con Salmonella y tratadas con fagos, como modelo de seguridad alimentaria (FOOD), y en terapia fágica oral en pollos de engorde contaminados con Salmonella (TF). Este estudio, publicado en la revista Frontiers in Cellular and Infectious Microbiology, evidencia que la frecuencia de aislamiento de dichas variantes en LAB es mucho mayor que la encontrada en FOOD y en TF.
Se demuestra que las causas de la emergencia de estas variantes son distintas en función del escenario. Así, en LAB y FOOD estas variantes son resistentes a fagos por mutaciones en los genes rfc y rfaJ implicados en la síntesis de los receptores fágicos. Sin embargo, no se han detectado variantes resistentes a fagos en TP, pero sí variantes que deben contener algún mecanismo de interferencia con el ciclo multiplicativo de los fagos. Dichos mecanismos deben estar codificados en plásmidos conjugativos presentes en la microbiota intestinal de los pollos. Así, se ha demostrado por vez primera que la adquisición de plásmidos por transferencia lateral puede tener mayor relevancia que la mutagénesis y también se ha constatado que la probabilidad de que ocurran estos eventos no afecta significativamente el éxito del biocontrol de alimentos y de la terapia fágica oral.
M. Pilar Cortés Garmendia
Departamento de Genética y de Microbiología
Universitat Autònoma de Barcelona
mariapilar.cortes@uab.cat
Montserrat Llagostera Casas
Departamento de Genética y de Microbiología
Universitat Autònoma de Barcelona
montserrat.llagostera@uab.cat
Referencias
López-Pérez J, Otero J, Sánchez-Osuna M, Erill I, Cortés P, Llagostera M. Impact of mutagenesis and lateral gene transfer processes in bacterial susceptibility to phage in food biocontrol and phage therapy. Front. Cell. Infect. Microbiol. (2023). Volume 13 - 2023 | doi: 10.3389/fcimb.2023.1266685


