Avaluada una nova tècnica ràpida per detectar tuberculosi resistent
La mortalitat per tuberculosi ja no només és un problema del tercer món. Les dades són clares: el nombre de casos en països desenvolupats augmenta progressivament. Si, a més a més, tenim en compte que el nombre de soques resistents als fàrmacs de primera línia també ha crescut, la situació pot arribar a ser greu. Per controlar adequadament la malaltia és necessari diagnosticar a les persones malaltes abans que l’afectat pugui infectar a una altra persona. Per aconseguir-ho, una de les possibilitats és la detecció de les mutacions responsables de les resistències al DNA del Mycobacterium tuberculosis -el microorganisme que causa la malaltia-, i el següent article ha avaluat l’eficàcia d'una tècnica molecular per a la detecció de resistències (
La tuberculosis continua matant a 2 milions de persones cada any. Malgrat que el problema és especialment greu en països en vies de desenvolupament, la incidència de la malaltia també està augmentant al nostre medi. L’augment de soques del bacteri resistents als fàrmacs de primera línia (més eficients i menys tòxics) agreugen la situació. El lent creixement de Mycobacterium tuberculosis, el bacil causant de la malaltia, fa que es retardi varies setmanes el resultat de l’estudi de susceptibilitat als antimicobacterians, de forma que si un malalt està infectat amb una soca multiresistent, el risc de que la transmeti i contagiï als seus contactes abans de que es detecti la situació, és molt elevat. Per a detectar precoçment aquestes soques, s’han desenvolupat diferents tècniques moleculars que detecten al DNA del bacil les principals mutacions que causen les resistències, de forma que no cal esperar al cultiu i la detecció de la resistència és molt més ràpida.
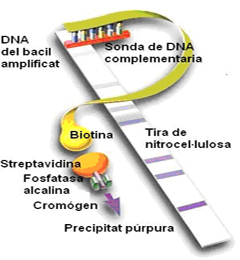
Exemples de possibles resultats i descripció de la sonda que hi ha fixada a cada banda.
Per aquest motiu, vam voler avaluar la utilitat d’una tècnica que combina una fase d’amplificació mitjançant PCR amb una tècnica de hibridació reversa en tires de nitrocel·lulosa (Genotype MTBDRplus) per a la detecció resistències a la rifampicina (mutacions al gen rpoB) i la isoniazida (mutacions als gens inhA i katG), tot comparant amb els resultats de detecció fenotípica convencional (BACTEC 460TB) i en el cas de les soques clíniques amb la seqüenciació. L’assaig es va avaluar en 62 soques clíniques de M. tuberculosis i en 65 mostres clíniques directes (39 amb resultat de examen microscòpic positiu i 26 amb resultat negatiu). La concordança global entre els resultats de MTBDRplus i BACTEC 460TB per a la detecció de susceptibilitat en soca clínica va ser de 98.3% per la rifampicina i de 79% per la isoniazida. Respecte les mostres clíniques directes, es van obtenir resultats vàlids al 78.45% de les mostres. En aquestes mostres, la concordança entre MTBDRplus i BACTEC 460 va ser de 98% per a la rifampicina i de 96.2% per a la isoniazida. La concordança es menor per a la Isoniazida degut a que les mutacions causants de la resistència en les soques que tenen baix nivell de resistència es troben en gens poc estudiats que no s’analitzen al test.
Amb els resultats obtinguts podem concloure que la tècnica avaluada és una eina útil i pràctica pel maneig de la tuberculosi, ja que permet la detecció ràpida de resistències als dos principal fàrmacs antituberculosos en soques i mostres clíniques directes.
Referències
GenoType MTBDRplus Assay for Molecular Detection of Rifampin and Isoniazid Resistance in Mycobacterium tuberculosis Strains and Clinical Samples. A. Lacoma, N. Garcia-Sierra, C. Prat, J. Ruiz-Manzano, L. Haba, S. Rosés, J. Maldonado, and J. Domínguez. JOURNAL OF CLINICAL MICROBIOLOGY, Nov. 2008, p. 3660?3667


