Una bacteria que puede ser un amigo peligroso
Las infecciones por Haemophilus parasuis producen importantes pérdidas económicas en el sector porcino. Aunque H. parasuis es una bacteria que forma parte de la microbiota normal del tracto respiratorio superior de los cerdos, algunas cepas también producen pneumonía y enfermedad sistémica, ya que su capacidad patógena les permite diseminarse desde el pulmón a otros órganos del animal. La variabilidad patológica de distintas cepas de esta bacteria se ha demostrado también en infecciones experimentales.
Como los factores de virulencia de H. parasuis no son conocidos, para diferenciar las cepas patógenas de las que no lo son, varios estudios han intentado relacionar la virulencia de las cepas con otras características, como el serotipo, el perfil de proteínas o el genotipo. En este sentido, los métodos genéticos presentan varias ventajas, ya que pueden permitir la clasificación de todas las cepas de una manera rápida e inequívoca. Así, se han desarrollado varios métodos genéticos para diferenciar las cepas de H. parasuis.
Sin embargo, las clasificaciones obtenidas hasta el momento no han sido totalmente satisfactorias. Además, en algunos casos los resultados de diferentes laboratorios son muy difíciles de comparar. Para eliminar este problema, en nuestro estudio usamos un método de secuenciación de fragmentos (aprox. 500 nucleótidos) de 7 genes conservados o “multilocus sequence typing” (MLST). Se estudiaron un total de 131 cepas de H. parasuis, que incluían 11 cepas de referencia de virulencia conocida y 120 cepas de campo de distintos orígenes, tanto clínicos como geográficos.
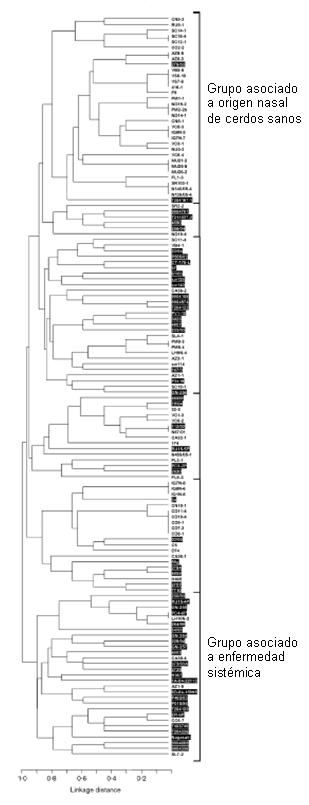
El análisis de los distintos alelos obtenidos produjo una clasificación de las cepas en 6 grupos. Cuando se analizó la asociación de estos grupos con los distintos orígenes de las cepas, se observó que un grupo estaba estadísticamente asociado a patología sistémica (cepas virulentas) y otro grupo a aislamiento de nariz de cerdos sanos (microbiota normal). El resto de los grupos no mostró una asociación significativa con el origen de las cepas.
Los resultados también indicaron que la recombinación es un fenómeno muy frecuente en estas cepas; es decir, intercambian material genético con facilidad. Este fenómeno ya lo habíamos observado anteriormente y tiene gran importancia cuando los genes transferidos están implicados en resistencia a antibióticos o virulencia. A pesar de la alta recombinanción, al comparar las secuencias concatenadas de los 7 genes, se detectaron dos subpoblaciones divergentes relacionadas con mayor o menor virulencia de las cepas. Una de estas subpoblaciones está claramente separada del resto e incluye cepas muy virulentas (asociadas a enfermedad invasiva).
Referencias
Alex Olvera, Marta Cerdà-Cuéllar, Virginia Aragón. Study of the population structure of Haemophilus parasuis by multilocus sequence typing. Microbiology (2006), 152: 3683-3690.


