Un nuevo mecanismo de regulación celular al descubierto
En esta época de crecimiento exponencial en el conocimiento de la regulación y de la composición genética de los seres vivos, y concretamente del funcionamiento de sus células, se ha llegado a la paradoja de la existencia de un bajo número de genes codificantes (aproximadamente unos 30.000 en humanos) a pesar de la gran diversidad de funciones celulares que existen. Esta gran diversidad de funciones celulares están realizadas por la acción de proteínas, y tan solo un número muy reducido de éstas están codificadas en nuestro genoma.
Una manera de diversificar las funciones proteicas en las células se realiza mediante las modificaciones post-traduccionales, en las cuales cambios producidos directamente sobre proteínas permiten modular su actividad dentro de las células. Las modificaciones post-traduccionales, denominadas así porque tienen lugar después de la traducción o síntesis proteica, son muy diversas: simples como las fosforilaciones (adición de un grupo fosfato), o más extensas como las glucosilaciones o lipidaciones (adición de cadenas de glúcidos o lípidos, respectivamente). En el grupo de investigación de los investigadores del IBB estudian la modificación post-traduccional por adición de otra proteína, denominada SUMO. Este proceso se conoce como Sumoilización.
SUMO es una proteína de bajo peso molecular (unos 80 aminoácidos) que es capaz de enlazarse a ciertas proteínas celulares a través de su extremo C-terminal formando un nuevo tipo de enlace llamado iso-peptídico. Actualmente se conocen más de 300 proteínas susceptibles a ser Sumoilizadas y que participan en importantes procesos celulares, desde la transcripción génica hasta mecanismos de reparación del material genético. Una de las principales características de esta modificación post-traduccional es que es reversible, existe una familia de proteínas, llamadas proteasas, que son capaces de revertir este proceso y liberar SUMO de la proteína que está modificando, cambiando de este modo nuevamente su actividad celular.
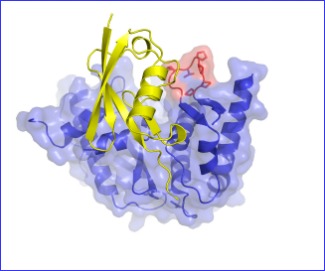
Los investigadores del IBB han descrito en su investigación los mecanismos que tienen que ver con la regulación del funcionamiento de estas proteasas de SUMO. Más concretamente han identificado una región en estas proteasas que es capaz de distinguir entre las dos variedades de SUMO que existen, SUMO1 y SUMO2. Esta propiedad permite a las proteasas de SUMO decidir si actúan liberando SUMO1 o SUMO2 de la proteína modificada y revertir su actividad. La capacidad de diferenciación de las dos variedades de SUMO por parte de esta familia de proteasas es nueva y es muy importante a la hora de activar o inhibir ciertos procesos que tienen lugar dentro de las células que están regulados por estas variedades.
En su estudio los investigadores han combinado estudios funcionales realizados en tubos de ensayos de modificación de proteínas por SUMO con estudios estructurales realizados por cristalografía de proteínas y de difracción de rayos X, utilizando la luz de sincrotrón.
Referencias
"Swapping Small Ubiquitin-like Modifier (SUMO) Isoform Specificity of SUMO Proteases SENP6 and SENP7".Alegre KO, Reverter D. J Biol Chem. 2011 Oct 14;286(41):36142-51.


